传统ChIP-seq与EMSA方法:实验周期长,需细胞培养/抗体开发;灵敏度有限,难以捕捉低表达或低亲和性结合。
结合Hdock与AlphaFold3高通量预测平台,三轮筛选构建核酸–TF互作复合体结构,揭示调控位点与结合机制。
(1)初筛(Hdock): 多模式对接 + 结合能评分,预选候选互作对(≤ -280 kcal/mol)。
(2)次筛(Hdock): 二轮能量优化,筛选TOP10结合模型。
(3)高精度建模(AF3): 构建复合体结构,验证结合界面可信度(pLDDT、iPTM)
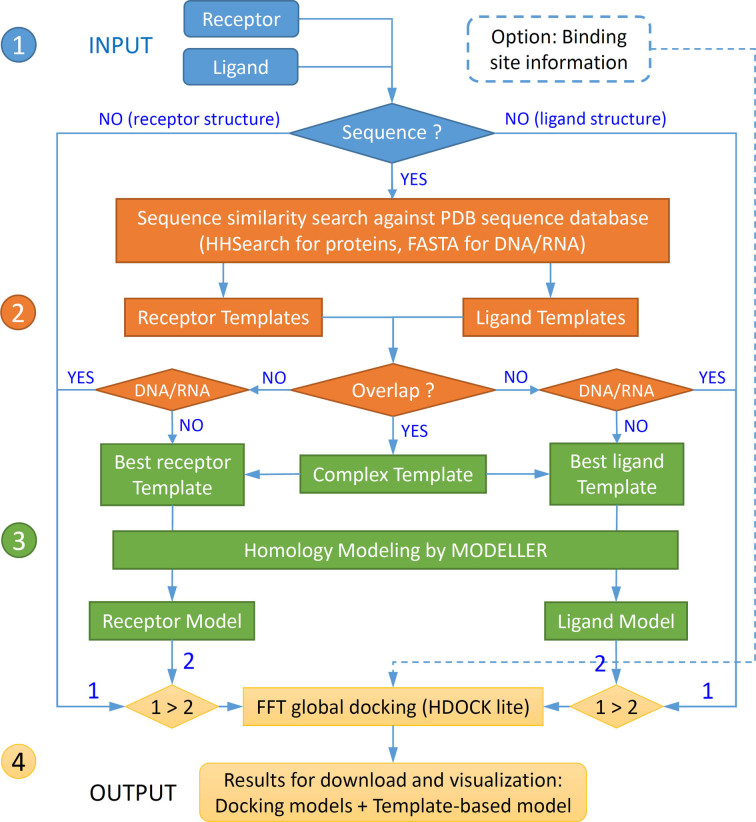
高效筛选 + 结构验证,周期缩短至 1–3 天
多模式对接,适配不同TF-DNA互作特性
结构结果可直接用于后续功能验证与分子设计
| 交付项 | 说明 |
| 候选互作对列表 | 包含预测的转录因子(TF)与核酸结合对,标注结合能(ΔG)、特异性评分(如P值)及置信度(pLDDT/iPTM)。 |
| 复合物结构模型 | 高精度TF-DNA/RNA复合物三维结构文件(AF3验证),支持可视化分析。 |
| 结合模式分析报告 | 详细描述结合界面关键残基、氢键/疏水作用网络及动态构象变化(如柔性对接轨迹)。 |
| 多维评分表 | 自由能(ΔG)、构象稳定性(RMSD)、结合特异性(Z-score)等量化指标汇总。 |
| 筛选流程文档 | 完整记录算法参数(Hdock/AlphaFold3)、筛选步骤、验证方法及潜在局限性说明。 |
[1] Abramson, J., Adler, J., Dunger, J. et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature 630, 493–500 (2024). https://doi.org/10.1038/s41586-024-07487-w
[2] Yan Y , Tao H , He J ,et al.The HDOCK server for integrated protein–protein docking[J].Nature Protocols, 2020, 15(Suppl 25):1-24.DOI:10.1038/s41596-020-0312-x.