许多分子作用过程无法通过静态结构解释:
结合构象变化剧烈、能量跃迁显著
动态通道/开关机制、膜嵌入行为等需要时间分辨分析
实验难以解析细节或观察稀有事件(如构象翻转、盐桥中断)
致一生物基于GROMACS模拟平台,结合AI建模、增强采样技术,支持蛋白、DNA/RNA、膜蛋白及复合体体系的高精度动态模拟。
| 组件 | 技术说明 |
| 力场体系支持 | CHARMM36、AMBER、OPLS-AA,多残基参数库 |
| 特殊残基支持 | 支持金属离子、磷酸化/甲基化/PEG修饰 |
| 增强采样 | REMD、副本交换、元动力学支持长尺度事件采样 |
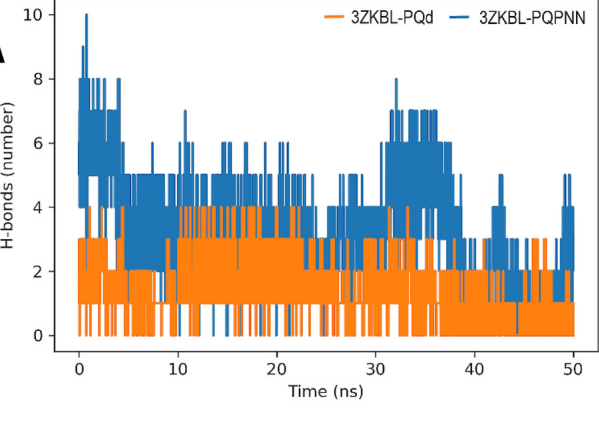
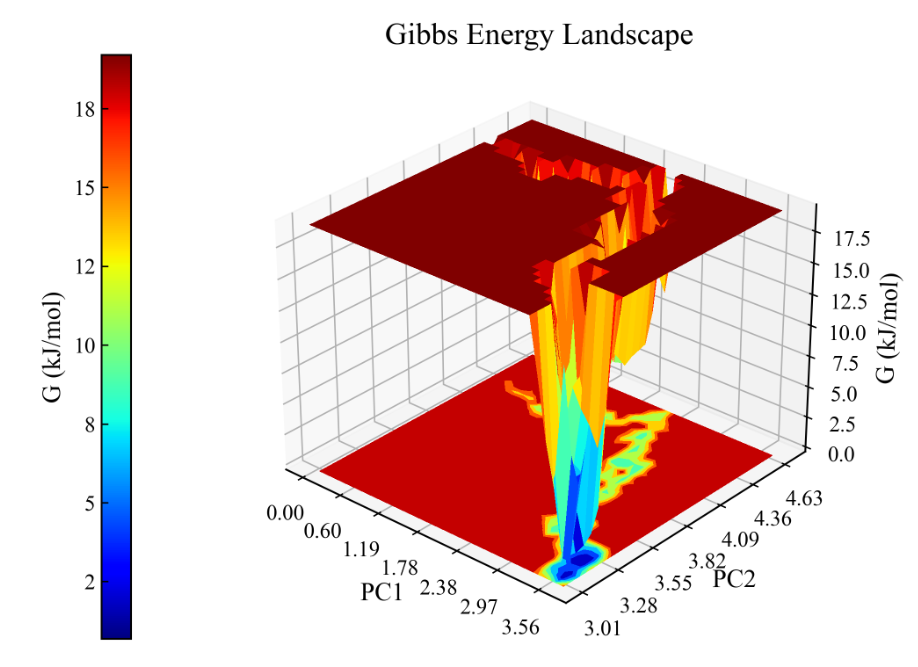
| 可视化与分析模块 | 提供RMSD、RMSF、SASA、能量曲线、Hbond等输出 |
结构准备与体系构建(蛋白/膜/复合体)
能量最小化 → 温度平衡 → 生产模拟(10ns–1μs)
分析阶段:轨迹提取 + 指标计算 + 图形输出
1、模拟轨迹文件(.xtc)与拓扑文件(.tpr、.gro)
2、分析图表(RMSD、自由能、氢键等)
3、可视化动画(mp4或pymol脚本)
4、报告包含参数设定与推荐后续实验设计
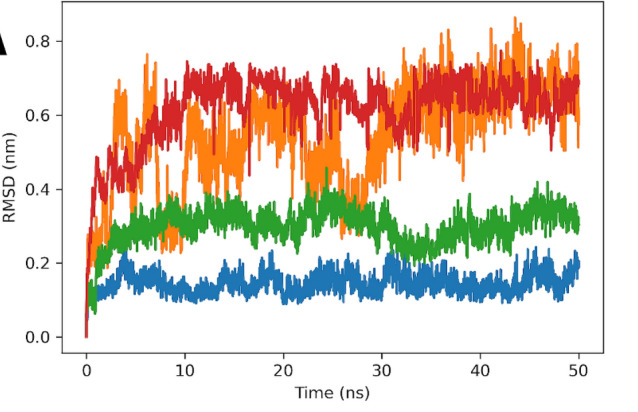
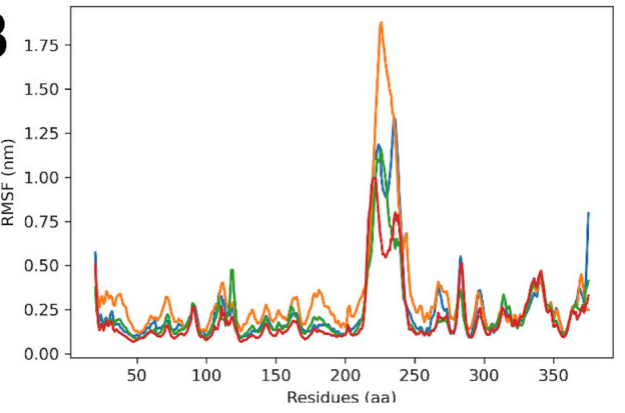
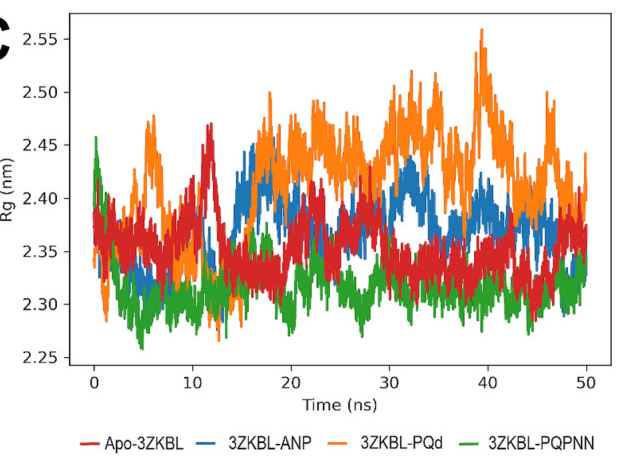
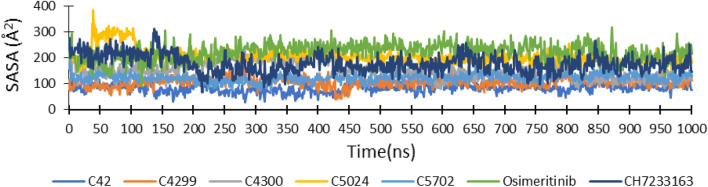
[1] Abraham, M. J. et al. GROMACS: High performance molecular simulations through multi-level parallelism. J. Chem. Theory Comput. 11(2), 542–555 (2015).